1ミリでいいからコロナに反撃したいエンジニアのための“仮想特効薬”の作り方:世界を「数字」で回してみよう(63) 番外編(4/7 ページ)
コピペするだけで、設計できてしまった……
それでは、早速江端さんにコロナウイルスを分解するsiRNAを設計していただきます。
まず、コロナウイルス(SARS-CoV-2)のゲノム情報を得ます。
こちらのサイトに、SARS-CoV-2のゲノム情報が公開されています。これは、武漢のオリジナルのウイルスのゲノム情報だったと思います。他にも変異株が続々に登録されていますが、まずは武漢のオリジナルで設計してみましょう。
ページの下の方、というか、一番下、にATGCの4文字が3万文字ほど並んだ配列があります。
これが憎っくきコロナウイルスのゲノムの全てです。
この1本鎖RNAをタンパク質と脂質の膜でくるんだらウイルスそのものになる、というわけです。
数字込みでいいので、originの表記の後、「1 attaa・・・・・」から、最後の「・・・・aaaaaa」の配列情報をコピーします。
(江端の脳内)
でも、3万塩基対から、クスリとして有用な塩基配列の候補を抽出って……どんなものすごい計算量になるんだろう。
私の作った交通シミュレーションでは、ゲーミングPC(高速処理に特化したPC)でも、合計1万人の人間を1日分移動させるだけで、3時間はかかったくらいだから ――
SARS-CoV-2を攻撃しつつ、他のRNAは攻撃しない ―― そんな都合の良い塩基対列を抽出するなら、最初の1つ目の塩基列発見に2〜3日くらいは必要だろうなぁ……。
次に、こちらのサイトを開きます。無料でsiRNAを設計してくれるたくさんあるホームページのうちの1つです。
え? 『無料』? 『たくさんあるホームページの1つ』? dry laboratoryの世界って、そんなに気前いいの?
枠内に、サンプル配列が記載されていますが、これを削除して、先ほどコピーした配列をペーストして、実行(クリック)してください。
はい。
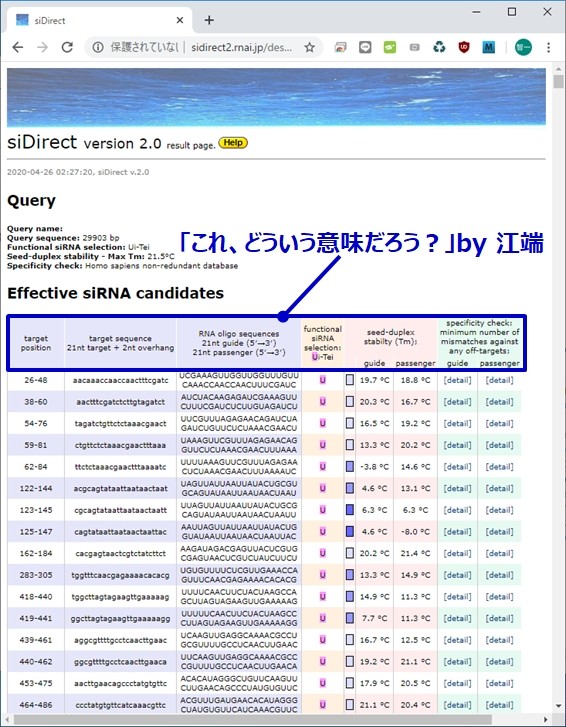
すると、大量のsiRNA配列候補が出力されてきます。
待つこと10秒 ―― すごいものが出てきました。
候補となるクスリ(の塩基配列) ―― 実に1809種類
*)ちなみに、今回使用したPCは、現在、秋葉原の中古PC店で19800円くらいで販売されている程度のPC(Intel Core i7-4770S CPU 3.10G、メモリ16GB)です。
おめでとうございます。抗SARS-CoV-2薬候補がたくさんデザインできました。
ありがとうございます……って、先生、そうじゃなくて(思わず、ノリツッコミをする江端)。
その瞬間、私の頭に浮んだことは、『医薬の設計製造は、高度な医療知識がある一部のエリートと、スパコンの仕事』という言い逃れが、もう使えなくなっているんだなぁ』という、隔世の感でした。
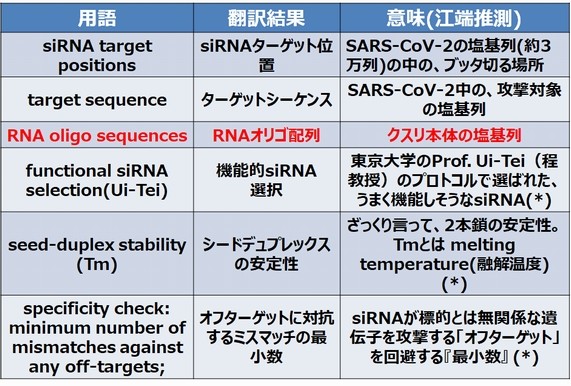
しかし、次の瞬間には、Webページに記載されている、江端が見たこともない用語(英語)を、Google翻訳に放り込み、自分なりに推測を始めていました。「まずは内容を理解せねば」と思ったからです。
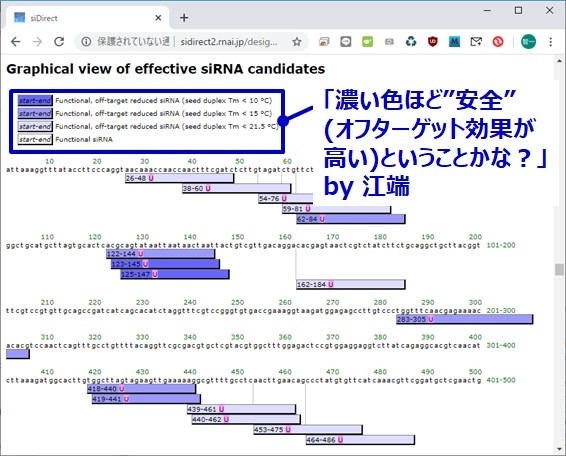
さらに、画面を下の方にスクロールしてみたら、候補となるクスリ(の塩基配列)が、図示されていました。
「いつの間にか、自宅のPCで、GUIを使って医薬を設計できる時代になっていたんだ」と、今さらながら、自分の”医薬リテラシー”の、あまりの低さに絶望してしまいました。
Copyright © ITmedia, Inc. All Rights Reserved.
記事ランキング
- ローム、TSMCのライセンス取得し650V GaNパワー半導体を自社生産へ
- 定年間際のエンジニアが博士課程進学を選んだ「本当の理由」
- Intelとソフトバンク子会社が次世代メモリ開発へ 29年度に実用化
- TIがSilicon Labsを75億ドルで買収する理由
- AIは「バブル」ではない――桁違いの計算量が半導体に地殻変動を起こす
- ソシオネクスト増収減益、中国向け車載新規品は順調に増加
- AI時代のニーズ捉え開発加速、キオクシア次期社長の展望
- SAIMEMORYの新構造メモリ 低消費電力に焦点
- 三菱電機の半導体は光デバイス好調 3Q受注高49%増
- TSMCは2nmで主導権維持、SamsungとIntelに勝機はあるか

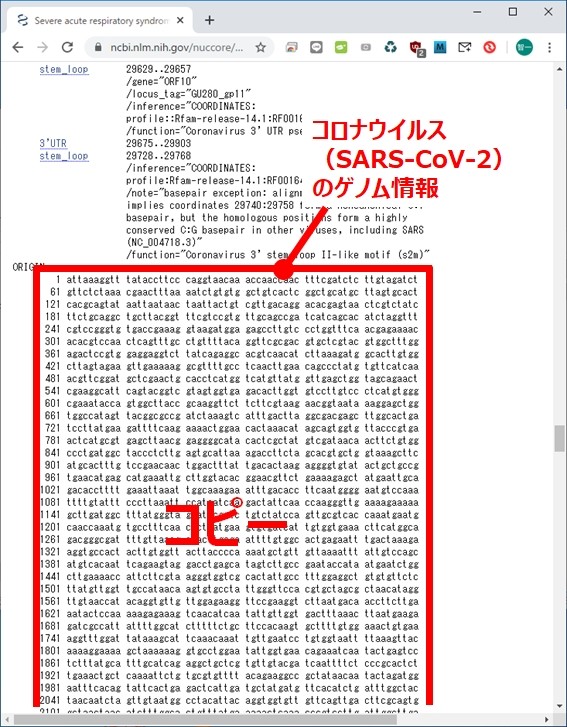 SARS-CoV-2のゲノム配列。Wordに、そのまんまコピーしたら14ページになりました。
SARS-CoV-2のゲノム配列。Wordに、そのまんまコピーしたら14ページになりました。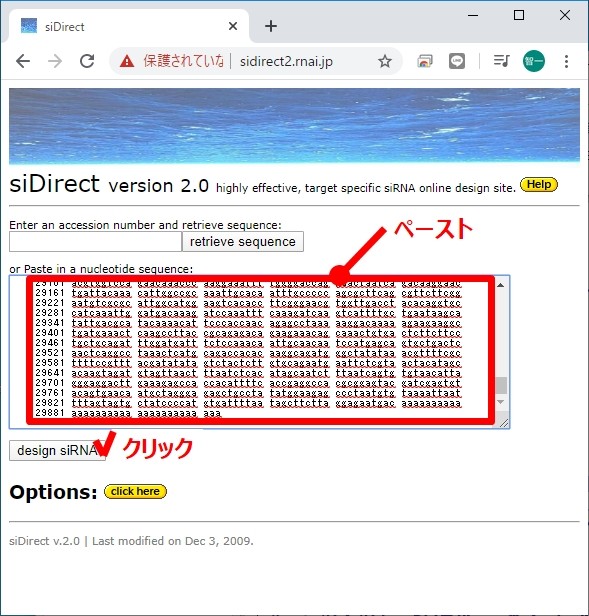
 (*):シバタ先生による詳細解説は
(*):シバタ先生による詳細解説は