感染症の解析に貢献するPCR検査と遺伝子検査、迅速検査(後編):福田昭のデバイス通信(388) 2022年度版実装技術ロードマップ(12)(2/2 ページ)
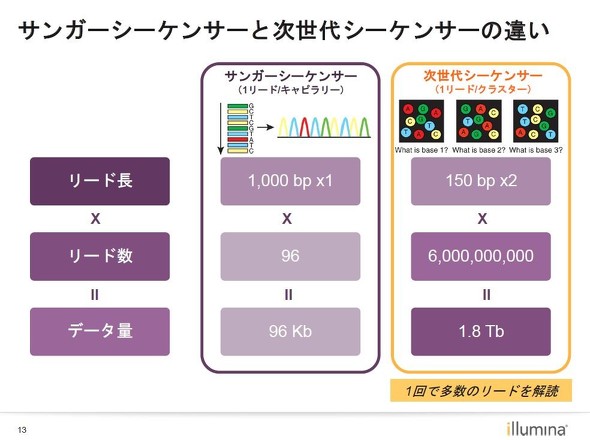
サンガー法と次世代シーケンス法の違い
1回のシーケンスで読み出せる塩基配列(データ)の数は、リード長(読み出すサンプルの塩基対の長さ、単位はbp(base pair))とリード数(一度に読み出すサンプルの数)の積によっておおむね決まる。サンガー法では、塩基対(bp)の数で1000を超える長さ(リード長)のDNAを扱える。ただし一度に読み出すサンプル数(リード数)は100未満と少ない。
これに対して次世代シーケンス(NGS)法は、リード長は数百bpと短いものの、リード数は1cm2当たりで数千万、総数(スライドガラスに搭載したサンプル)では数十億に達する。例えばNGSの代表的な開発企業であるilluminaの資料によると、1回のサンガー法が読み出すデータ量は96Kbp(9万6000塩基)であるのに対し、1回のNGSではサンガー法の1875万倍、1.8Tbp(1兆8000億塩基)ときわめて膨大な数の塩基配列を解読できる。
また最近では、リード長を10Kbpから100Kbpに伸ばしたNGSが製品化されている。このタイプのNGSを「ロングリードNGS」と呼び、従来のNGSを「ショートリードNGS」と呼んで区別することがある。
「腸内フローラ」の研究を活発化させた遺伝子解析の高速化と低コスト化
次世代シーケンス(NGS)法の高速化と低コスト化は、遺伝子検査の応用分野を大幅に拡大した。中でも「腸内細菌叢(ちょうないさいきんそう)(「腸内フローラ」、「腸内バイオーム」とも呼ばれる)」の研究は、遺伝子解析技術によって近年、急速に活発化してきた。例えば2018年に発表された腸内細菌叢の学術論文は5000件を超えており、この数は10年前の10倍を超えるという(参考文献:井上、「腸内細菌叢解析のいろは」、『日本乳酸菌学会誌』、2019年3月号、p.27)。
腸内細菌叢に限らずヒトの細菌叢(細菌の集団)は、数(種類および総数)が多すぎる、培養には一定の技能を必要とする、培養困難な細菌が少なくないといった理由で、研究があまり進んでいなかった。特に大腸は管内に酵素が存在せず、実験室における細菌の培養を困難にしていた。ところが前述のように培養を必要としない、次世代シーケンス技術を活用した細菌叢の遺伝子解析が近年は非常に盛んになった。
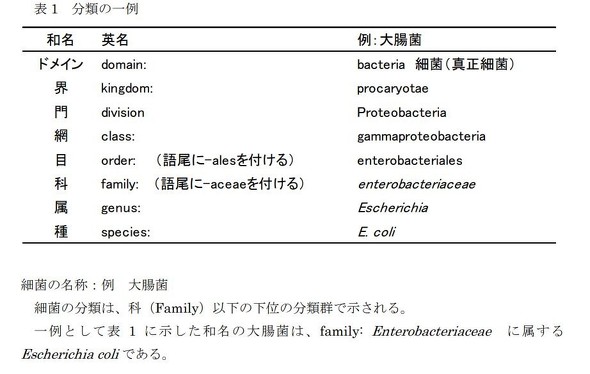
腸内細菌叢を例として取り上げると、遺伝子解析には「アンプリコン解析」と「メタゲノム解析」の2つの手法が使われている。「アンプリコン解析」とはDNAに含まれる特定の遺伝子領域だけをPCR増幅して解析する手法である。腸内細菌叢のアンプリコン解析には、「16S rRNA」と呼ぶ遺伝子を利用する「16S rRNA遺伝子解析」を使うことが多い。細菌叢全体のDNAから16S rRNA遺伝子をPCR増幅してサンプルDNAを精製し、次世代シーケンス装置を使って塩基配列を読み取る。
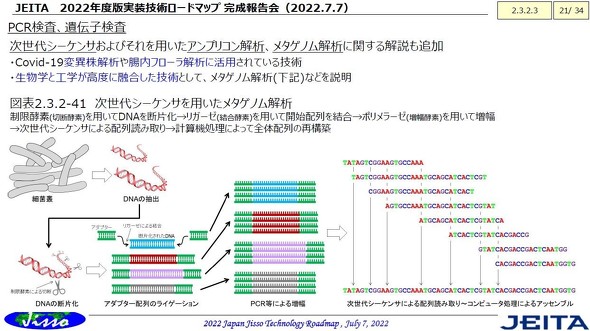 2022年度版ロードマップでは、次世代シーケンサー、アンプリコン解析、メタゲノム解析に関する記述を追加した。下の図はメタゲノム解析の主な手順。出所:JEITA Jisso技術ロードマップ専門委員会(2022年7月7日に開催された完成報告会のスライド)
2022年度版ロードマップでは、次世代シーケンサー、アンプリコン解析、メタゲノム解析に関する記述を追加した。下の図はメタゲノム解析の主な手順。出所:JEITA Jisso技術ロードマップ専門委員会(2022年7月7日に開催された完成報告会のスライド)「16S rRNA遺伝子解析」では、細菌を「門」や「科」、「属」などで分類したグループの占有率が得られる。解析コストは低いものの、詳細な解析は難しい。
解析コストは高くなるものの、詳細な解析を可能にするのが「メタゲノム解析」である。細菌叢のDNAを抽出して断片化し、PCR増幅によってDNA断片の数を増やし、次世代シーケンス技術によって塩基配列を網羅的に読み取る。読み取った塩基配列(DNA断片)をコンピューター処理によって接続し、DNA全体の塩基配列を再構築する。
関連記事
 感染症の解析に貢献するPCR検査と遺伝子検査、迅速検査(前編)
感染症の解析に貢献するPCR検査と遺伝子検査、迅速検査(前編)
今回と次回は、JEITAの「2022年度版 実装ロードマップ」から、「感染症とPCR検査、遺伝子検査、迅速検査」(2.3.2.3)の概要をご紹介する。 メディカル分野の課題解決に貢献する実装技術
メディカル分野の課題解決に貢献する実装技術
今回からは第2項(2.3.2)「メディカル」の概要を報告していく。「メディカル」は「手術支援ロボット」(2.3.2.1)、「マイクロ流体デバイス」(2.3.2.2)、「感染症とPCR検査、遺伝子検査、迅速検査」(2.3.2.3)、「バイオセンサ」(2.3.2.4)の4つの項目で構成される。 実験室の機能を数cmサイズに集積したマイクロ流体デバイス
実験室の機能を数cmサイズに集積したマイクロ流体デバイス
JEITAの「2022年度版 実装ロードマップ」を紹介するシリーズ。今回は、第2章「注目される市場と電子機器群」から「マイクロ流体デバイス」の概要を簡単にご紹介する。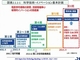 日本政府の科学技術政策と実装技術ロードマップの重要性
日本政府の科学技術政策と実装技術ロードマップの重要性
今回から、「2022年度版 実装ロードマップ」の第2章「注目される市場と電子機器群」の概要を複数回に分けて説明していく。 3年ぶりに更新されたエレクトロニクスと実装技術の将来展望
3年ぶりに更新されたエレクトロニクスと実装技術の将来展望
今回から「2022年度版 実装技術ロードマップ」の概要を連載シリーズでご報告する。 オンチップの相互接続技術を過去から将来まで概観
オンチップの相互接続技術を過去から将来まで概観
2020年6月にオンラインで開催された「VLSIシンポジウム」から、オンチップの多層配線技術に関するIntelの講演内容を紹介する。
Copyright © ITmedia, Inc. All Rights Reserved.
記事ランキング
- ローム、TSMCのライセンス取得し650V GaNパワー半導体を自社生産へ
- 定年間際のエンジニアが博士課程進学を選んだ「本当の理由」
- Intelとソフトバンク子会社が次世代メモリ開発へ 29年度に実用化
- AIは「バブル」ではない――桁違いの計算量が半導体に地殻変動を起こす
- TIがSilicon Labsを75億ドルで買収する理由
- AI時代のニーズ捉え開発加速、キオクシア次期社長の展望
- ソシオネクスト増収減益、中国向け車載新規品は順調に増加
- SAIMEMORYの新構造メモリ 低消費電力に焦点
- 三菱電機の半導体は光デバイス好調 3Q受注高49%増
- TSMCは2nmで主導権維持、SamsungとIntelに勝機はあるか